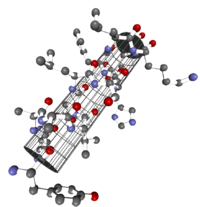
α-hélice
En biochimie, l' hélice α est une expression courante de la structure secondaire d' une protéine . C'est l'une des conformations naturelles les plus stables d' une séquence d'acides aminés et est presque omniprésente dans la structure secondaire.
Par structure secondaire d'une protéine, on entend la structure spatiale de la chaîne d'acides aminés sans tenir compte des groupes latéraux. La structure secondaire d'une protéine est dérivée de sa structure primaire (séquence d'acides aminés). Les niveaux structurels supérieurs sont la structure tertiaire et la structure quaternaire . La structure tridimensionnelle d'une protéine est cruciale pour sa fonction sélective (voir structure de la protéine ).
l'histoire
À la fin des années 1930, William Astbury a commencé à effectuer des analyses de la structure cristalline sur des peptides cristallins . Il a été constaté que certaines caractéristiques spatiales se répètent régulièrement dans lesquelles des liaisons hydrogène au sein de la molécule étaient suspectées. Cependant, il n'était pas encore conscient de la planarité de la liaison peptidique . Les structures spatiales les plus courantes ont été appelées plus tard hélice α et feuillet β . Linus Pauling , Robert Brainard Corey et Herman Branson ont proposé un modèle de l'hélice α en 1951. Le α dans "α-hélice" ne contient aucune déclaration scientifique, mais exprime seulement le fait que α-hélice a été trouvée devant la feuille β. Le tracé de Ramachandran développé par G. N. Ramachandran a permis de les identifier en utilisant les angles dièdres des acides aminés consécutifs dans la protéine , similaire au tracé de Janin développé plus tard .
structure
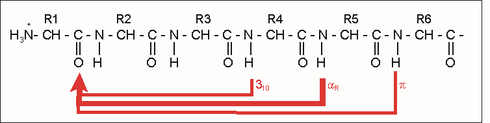
L'hélice a est une spirale droite (de préférence de L-acides aminés) avec une moyenne de 3,6 chaînes latérales d'acides aminés par révolution. Une longueur de p = 0,54 nm (5,4 Â) est obtenue par tour. Cette avance est connue sous le nom de pitch. C'est le produit du décalage (également appelé translation) (0,15 nm) et des restes par tour (3,6). Cette distance entre les résidus est la raison pour laquelle les acides aminés séparés de trois ou quatre endroits dans la structure primaire sont à proximité étroite dans la structure en hélice. L'hélice α est stabilisée par une liaison hydrogène entre l'oxygène carbonyle du nième et le proton amide du (n + 3) ième acide aminé de la même molécule.
Les groupes CO et NH doivent être proches l'un de l'autre pour former la liaison hydrogène. La configuration la plus étroite est fournie par un brin enroulé dans lequel les deux groupes se superposent. Les chaînes latérales pointent vers l'extérieur. L'acide aminé proline («structure breaker») ne peut pas être facilement inséré dans l'hélice (cela n'est possible qu'aux positions 1 à 4, vu de l'extrémité amino). En conséquence, il y a des écarts par rapport à la structure régulière aux points où la proline se produit. Les α-hélices sont très stables et, en tant que cylindres rigides, peuvent former une sorte de squelette de la protéine. Par conséquent, ils ne sont souvent pas représentés comme des hélices mais comme des cylindres dans les structures protéiques. Une protéine avec une structure principalement hélicoïdale est la myoglobine , une protéine musculaire liée à l' hémoglobine .
Une hélice α n'est souvent stable que dans le contexte d'une protéine, c'est pourquoi des liaisons stabilisantes supplémentaires sont souvent introduites dans des hélices α isolées, par ex. B. en remplaçant la liaison hydrogène par une liaison CC, en réticulant les chaînes latérales d'acides aminés ou en formant des ponts disulfure .
Géométrie des interactions hélice et hélice-hélice


La partie A montre une «bobine enroulée» composée de deux, la partie B une constituée de trois hélices α (projection). De plus, des structures de «faisceau tétrahélix» ont été décrites.

Les α-hélices sont à la base des protéines typiques des fibres (α-kératine, la substance de base des cheveux, myosine, un composant des fibres musculaires, etc.) mais aussi, comme introduit dans l'exemple de la myoglobine, des composants structurants des solubles, protéines globulaires. En général, les hélices individuelles ne peuvent pas assumer cette tâche, mais des agrégats ordonnés constitués de deux, trois, quatre hélices individuelles ou plus le peuvent.
L'assemblage pour former une telle «superhélice» est basé sur des interactions hydrophobes en hélices amphipathiques . Ce sont des hélices dont une face est hydrophile (face à l'eau) et l'autre est hydrophobe et donc susceptible d'interactions. La structure de l'hélice signifie que la «bande hydrophobe» ne s'étend pas parallèlement à l'axe de l'hélice, mais entoure l'hélice sous la forme d'une spirale étirée à gauche. Lorsque les bandes hydrophobes de deux ou plusieurs hélices se rapprochent, la superhélice connue sous le nom de « bobine enroulée » est formée.
Prédiction Helix
Les efforts initiaux pour prédire les structures secondaires des protéines remontent aux années 1960 et ont été continuellement affinés avec l'avènement de l' analyse moderne des structures aux rayons X. Une approche extrêmement utile et rationnelle pour prédire l'hélice α est associée au nom de Marianne Schiffer et fait suite aux considérations ci-dessus. Selon le critère n +/- 3,4, un reste n peut s'apparier à des résidus éloignés de trois ou quatre positions. Sont z. Si, par exemple, les résidus 1, 4 et 5 sont hydrophobes, ils peuvent interagir et ainsi stabiliser une structure en hélice. Il en est de même pour les résidus 6, 3 et 2 etc. Ce schéma de prédiction a initialement montré sa valeur pour l'insuline et la myoglobine.
Avec la publication de nouvelles analyses de structure aux rayons X, l'approche «roue hélicoïdale» a de plus en plus cédé la place aux méthodes statistiques. Une première approche de ce type remonte à Chou et Fasman (1974, 1978).
Le tableau ci-dessous montre les potentiels hélicoïdaux (Pα) des résidus d'acides aminés. Pα correspond à la fréquence relative avec laquelle l'acide aminé est représenté dans l'hélice. Avec un Pα bien au-dessus de 1, un acide aminé est appelé "formateur d'hélice", avec un Pα bien inférieur à 1 en tant que "casseur d'hélice".
| acide aminé | Pα |
|---|---|
| Glu | 1,59 |
| Ala | 1,41 |
| Leu | 1,34 |
| Hydromel | 1,30 |
| Gln | 1,27 |
| Lys | 1,23 |
| Arg | 1,21 |
| Phe | 1,16 |
| Ile | 1,09 |
| Le sien | 1,05 |
| Trp | 1,02 |
| Aspic | 0,99 |
| Val | 0,90 |
| Thr | 0,76 |
| Asn | 0,76 |
| Cys | 0,66 |
| Tyr | 0,61 |
| Ser | 0,57 |
| Gly | 0,43 |
| Par | 0,34 |
Résumé des paramètres d'hélice
| A - Relation des restes entre eux | |||
| n + 4 | Restes 1 et 5 | Pont en H | -C = O ··· HN- |
| n +/- 3, 4 | Restes 1 et 4 ou 5 | même côté | "Arc hydrophobe", potentiel α |
| n + 18 | Vestiges 1 et 19 | écliptique | 5 x 3,6 = 18; "Répéter l'unité" |
| n + 7 | Restes 1 et 8 | "Presque écliptique" | 2 x 3,6; "Répéter Heptad" |
| B - paramètres physiques | |||
| n = 3,6 | Restes par tour | ||
| d = 1,5 A | décalage axial par reste | ||
| p = 5,4 Å = nxd | "Pitch" (distance entre les bobines) | ||
| a = 100 ° = 360 ° / n | Angle (secteur) par acide aminé | ||
Autres formes de structure secondaire

En plus de l'hélice α et de la feuille β, il existe d'autres types de structure secondaire (motifs de structure secondaire). D'autres motifs communs sont:
Les parties de la structure primaire d'une protéine qui n'appartiennent pas à un motif sont appelées structures à bobines aléatoires . Ces structures sont également impliquées de manière significative dans la formation de toute la structure protéique .
Littérature
- M. Schiffer, AB Edmundson: Utilisation de roues hélicoïdales pour représenter les structures de protéines et pour identifier des segments à potentiel hélicoïdal. Dans: Biophysical Journal. 7, 1967, pages 121-135.
- PY Chou, GD Fasman: Prédictions empiriques de la conformation des protéines. Dans: Revue annuelle de biochimie . 47, 1978, pages 251-276.
- C. Cohen, DAD Parry: bobines hélicoïdales α - un motif répandu dans les protéines. Dans: Tendances des sciences biochimiques . 11, 1986, pages 245-248.
- S. Kamtekar, JM Schiffer, H. Xiong, JM Babik, MH Hechtr: Conception de protéines par structuration binaire d'acides aminés polaires et non polaires. Dans: Science . 262, 1993, pages 1680-1685.
liens web
Preuve individuelle
- ^ William T. Astbury, S. Dickinson, K. Bailey: L'interprétation aux rayons X de la dénaturation et la structure des globulines des graines. Dans: The Biochemical Journal. Volume 29, numéro 10, octobre 1935, p. 2351-2360.1, PMID 16745914 . PMC 1266766 (texte intégral gratuit).
- ^ William T. Astbury: Les protéines structurales de la cellule. Dans: The Biochemical Journal. Volume 39, numéro 5, 1945, p. Lvi, PMID 21020817 .
- ^ WT Astbury, R. Reed, LC Spark: Une étude au microscope à rayons X et électronique de la tropomyosine. Dans: The Biochemical Journal. Volume 43, numéro 2, 1948, p. 282-287, PMID 16748402 . PMC 1274681 (texte intégral gratuit).
- ^ Linus Pauling, Robert Brainard Corey, Herman R. Branson: La structure des protéines; deux configurations hélicoïdales liées à l'hydrogène de la chaîne polypeptidique. Dans: Actes de la National Academy of Sciences . Volume 37, numéro 4, avril 1951, p. 205-211, PMID 14816373 . PMC 1063337 (texte intégral gratuit).
- ↑ JM Scholtz, RL Baldwin: Le mécanisme de formation de l'hélice alpha par les peptides. Dans: Revue annuelle de la biophysique et de la structure biomoléculaire. Volume 21, 1992, pages 95-118, doi: 10.1146 / annurev.bb.21.060192.000523 . PMID 1525475 . rbaldwin.stanford.edu (PDF)
- ↑ Jeremy M. Berg, John L. Tymoczko, Lubert Stryer: Stryer Biochemistry . 7e édition. Springer Spectrum, Heidelberg 2014, ISBN 978-3-8274-2988-9 , p. 40 .
- ↑ A. Winter, AP Higueruelo, M. Marsh, A. Sigurdardottir, WR Pitt, TL Blundell: Approches biophysiques et basées sur des fragments informatiques pour cibler les interactions protéine-protéine: applications dans la découverte de médicaments guidés par la structure. Dans: Revues trimestrielles de la biophysique. Volume 45, numéro 4, novembre 2012, p. 383-426, doi: 10.1017 / S0033583512000108 . PMID 22971516 .
- ↑ LK Henchey, AL Jochim, PS Arora: Stratégies contemporaines pour la stabilisation des peptides dans la conformation alpha-hélicoïdale. In: Opinion actuelle en biologie chimique. Volume 12, numéro 6, décembre 2008, p. 692-697, doi: 10.1016 / j.cbpa.2008.08.019 . PMID 18793750 . PMC 2650020 (texte intégral gratuit).
- ↑ RJ Platt, TS Han, BR Green, MD Smith, J. Skalicky, P. Gruszczynski, HS White, B.Olivera, G.Bulaj, J.Gajewiak: L'agrafage imite les interactions non covalentes des γ-carboxyglutamates dans les conantokines, antagonistes peptidiques de Récepteurs de l'acide N-méthyl-D-aspartique. Dans: Le Journal de la chimie biologique. Volume 287, numéro 24, juin 2012, p. 20727-20736, doi: 10.1074 / jbc.M112.350462 . PMID 22518838 . PMC 3370255 (texte intégral gratuit).
- ↑ P. Barthe, S. Rochette, C. Vita, C. Roumestand: Synthèse et structure de la solution RMN d'une épingle à cheveux alpha-hélicoïdale agrafée avec deux ponts disulfure. Dans: Protein science: une publication de la Protein Society. Volume 9, numéro 5, mai 2000, p. 942-955, doi: 10.1110 / ps.9.5.942 . PMID 10850804 . PMC 2144636 (texte intégral gratuit).
- ↑ Jeremy M. Berg: Stryer Biochemistry. 6e édition. Spectrum Academic Publishing House, Heidelberg 2007, p. 56.
- ^ Focus sur la communauté PDB: Julian Voss-Andreae, sculpteur de protéines. Dans: Bulletin de la banque de données sur les protéines. 32, hiver 2007, wwpdb.org (PDF)
- ↑ L. Moran, RA Horton, G. Scrimgeour, M. Perry: Principes de biochimie . Pearson, Boston MA 2011, ISBN 978-0-321-70733-8 , p. 127 .