ADN polymérases
| ADN polymérase | ||
|---|---|---|

| ||
| Représentation en ruban du domaine de liaison à l'ADN de l'ADN polymérase humaine β | ||
| Identifiant | ||
| Identifiants externes | ||
| Classification enzymatique | ||
| CE, catégorie | 2.7.7.7 , Cession | |
| Substrat | Désoxyribonucléoside triphosphate + ADN n | |
| Des produits | Diphosphate + ADN n + 1 | |
Les ADN polymérases sont des enzymes qui, en tant que polymérases, catalysent la synthèse d' ADN à partir de désoxyribonucléotides . Les ADN polymérases jouent un rôle clé dans la réplication de l'ADN .
Aspects biochimiques
Activité polymérase
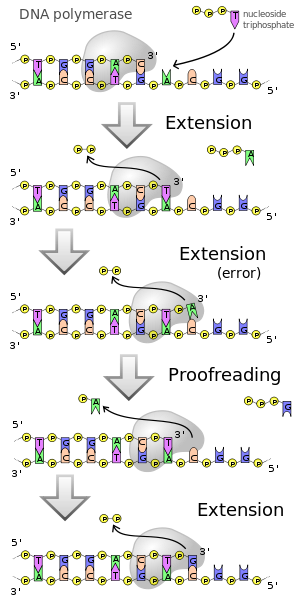
La polymérase permet la liaison chimique de molécules individuelles ( monomères ) à une chaîne ( polymère ). Dans le cas de l'ADN polymérase, le polymère formé est l'acide désoxyribonucléique (ADN) ; les désoxyribonucléotides , plus précisément les désoxy nucléoside triphosphates (dNTP), servent de monomères . L'ADN polymérase dépendante de l'ADN utilise toujours un simple brin d'ADN existant comme matrice (template) pour la synthèse d'un nouveau brin complémentaire, dont la séquence nucléotidique est ainsi déterminée par la matrice. Cette préservation de la séquence d'ADN est cruciale pour la capacité de l'ADN polymérase à copier l' information génétique codée dans l'ADN . La copie correcte de la matrice est obtenue grâce à l' appariement de bases complémentaires des bases nucléotidiques incorporées avec les bases de la matrice d'ADN, médiée par des liaisons hydrogène . La synthèse du nouveau brin d'ADN s'effectue de l' extrémité 5' à l'extrémité 3' . D'un point de vue chimique, le groupe terminal 3'- hydroxyle du brin d'ADN subit une attaque nucléophile sur le -phosphate du dNTP, libérant du pyrophosphate . Cette étape est catalysée par la polymérase.
Contrairement aux ARN polymérases , le brin d'ADN complémentaire ne peut être synthétisé dans les ADN polymérases que si la polymérase a une extrémité 3'-hydroxy libre disponible. Le premier nucléotide est alors attaché à celui-ci. Dans la réaction en chaîne par polymérase (PCR), un seul brin d'ADN d'environ 15 à 20 nucléotides de long ( amorce ) est utilisé, qui sert de point de départ à la réaction. Les ADN polymérases nécessitent généralement des ions magnésium comme cofacteur .
La catalyse de la formation de la liaison diester est fonctionnellement analogue à la réaction correspondante des ARN polymérases. Le dernier nucléotide de la section déjà synthétisée et le nucléotide à ajouter sont chacun coordonnés à l'un des deux ions magnésium dans le centre catalytique du domaine polymérase. Le premier groupe phosphate du nucléotide à ajouter est coordonné aux deux ions magnésium. La position spatiale permet au groupe hydroxyle du nucléotide précédent d' attaquer le groupe phosphate de celui à ajouter. Un résidu de pyrophosphate est séparé dans le processus.
Activité exonucléase
De nombreuses polymérases ont également d'autres fonctions enzymatiques. En présence de faibles concentrations de dNTP, l' activité exonucléase 3' → 5' d'élimination des nucléotides prédomine . Certaines polymérases ont également une activité exonucléase 5'→3' . Pour s'assurer qu'il n'y a pas d'erreurs dans la lecture de la matrice d'ADN, ils ont cette fonction de relecture (Engl. Proofreading ), c'est-à-dire qu'il peut être vu un nucléotide inapproprié capable de monter et ceci est par la suite en supprimant à nouveau l'activité exonucléase de l'ADN. Cela permet à un brin d'ADN ou d'ARN existant qui est déjà apparié avec le brin matrice d'être décomposé pendant la formation d'un nouveau brin. Le résultat est un échange de l'ancienne ligne contre une nouvelle ligne. Cette activité exonucléase est utilisée dans la méthode de translation de coupure .
Diverses ADN polymérases
Il existe trois ADN polymérases dépendantes de l'ADN différentes dans les bactéries telles que Escherichia coli . L'une d'entre elles, l' ADN polymérase I (Pol I), a été isolée par Arthur Kornberg en 1955 et a été la première polymérase jamais découverte. Cependant, ce n'est pas la polymérase la plus importante dans E. coli pour la réplication , car elle ne catalyse qu'environ 20 étapes de synthèse (c'est-à-dire qu'elle n'a qu'un faible niveau de processivité ). Cependant, en raison de son activité exonucléase 5' → 3', il est responsable de la dégradation des amorces lors de la réplication. L'ADN polymérase II et l' ADN polymérase III , les deux autres ADN polymérases dans E. coli , n'ont été isolées que 15 ans après la découverte de l'ADN polymérase I, après que des mutants d' E. coli avec un défaut dans le gène de la polymérase I se soient avérés être prouvés. pouvoir se répliquer . Cependant, ces mutants étaient particulièrement sensibles aux rayonnements UV et aux substances alkylantes, c'est pourquoi on suppose que l'ADN polymérase I assume principalement des tâches de réparation. La polymérase III, qui effectue la réplication proprement dite dans E. coli , est constituée d'un total de sept sous-unités et ne se produit qu'en très peu de copies par cellule bactérienne.
Les ADN polymérases eucaryotes sont classées dans les familles suivantes :
- Famille A : ADN polymérases γ, θ et ν
- Famille B : ADN polymérases α, δ, ε et ζ
- Famille X : ADN polymérases β, λ, σ et μ
- Famille Y : ADN polymérases η, ι et κ
La polymérase se produit uniquement dans les mitochondries .
Il n'y a que cinq ADN polymérases trouvées chez les mammifères : α, β, , et ε. On suppose que les polymérases δ et , qui sont décisives pour la réplication, sont caractérisées par une processivité et une relecture élevées . Les polymérases et , en revanche, ne présentent qu'une faible processivité et aucune fonction de relecture.
Il existe également des ADN polymérases dépendantes de l'ARN qui utilisent l'ARN comme matrice et y attachent des dNTP. Celles-ci sont appelées transcriptase inverse , qui comprend également la télomérase . En tant qu'ADN polymérase indépendante comme la seule qui est le désoxyribonucléotidyle terminal connu.
Chez les archaebactéries, il existe des ADN polymérases thermostables qui sont également utilisées pour la PCR .
Non 3 '→ 5' polymérases
Le traitement des complexes d'ADN polymérase en 3 '→ 5' n'est pas connu. Une extension du brin d'ADN dans cette direction nécessiterait l'hydrolyse du nucléoside triphosphate préalablement ajouté. Bien que cela soit possible en principe, cela pose un problème au niveau d'une fonction de correction supplémentaire ( relecture ). Car après la réaction d'une exonucléase, il n'y aurait pas de groupement triphosphate au bout du brin, mais seulement un simple groupement phosphate, ce qui empêcherait un allongement supplémentaire du brin. Le mécanisme de réaction hypothétique suivant peut illustrer cela :
Importance biologique
Les ADN polymérases sont au cœur de la réplication de l'ADN . Ils permettent la copie fidèle de l'information génétique sous forme d'ADN, donc une étape décisive dans la reproduction et la reproduction des êtres vivants. Les ADN polymérases jouent également un rôle important dans les processus associés à la réparation de l'ADN.
Importance biotechnologique
En laboratoire, les ADN polymérases sont souvent utilisées pour la réaction en chaîne par polymérase et les méthodes associées (par exemple RT-PCR , qPCR ), pour la translation de coupure , pour l'amorçage aléatoire et pour le séquençage de l'ADN . Un grand nombre d' ADN polymérases thermostables différentes, dont certaines ont été modifiées par génie protéique , sont utilisées (par exemple, la Taq polymérase ). En plus d'une stabilité à haute température, les ADN polymérases thermostables d'origine archaïque telles que la Pfu polymérase ont une fonction de relecture , puisque l'ADN produit ne doit pas être modifié lors de la PCR. En outre, les ADN polymérases à dislocation de brins telles que l' ADN polymérase φ29 sont utilisées dans diverses méthodes d' amplification d'ADN isotherme à température ambiante . Le précurseur des ADN polymérases utilisées aujourd'hui était l' ADN polymérase T4 .
Littérature
- Lehninger ; David Nelson, Michael Cox : Biochimie de Lehninger. 3e édition, Springer-Verlag, Berlin Heidelberg 2001, ISBN 3-540-41813-X
- Wilhelm Seyffert : Manuel de génétique. 2e édition, Spektrum Akademischer Verlag Heidelberg, Berlin 2003, ISBN 3-8274-1022-3
- Jeremy M. Berg, John L. Tymoczko, Lubert Stryer : Biochimie 6e édition, Springer-Verlag, Heidelberg 2007, ISBN 978-3-8274-1800-5
liens web
- Molécule PDB du mois : ADN polymérase
Preuve individuelle
- ↑ Donald Voet : Biochimie . 4e édition édition. John Wiley & Sons, Hoboken, NJ 2011, ISBN 978-0-470-57095-1 , p. 1201 .